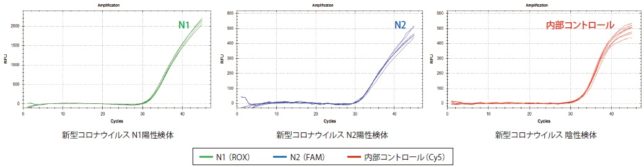
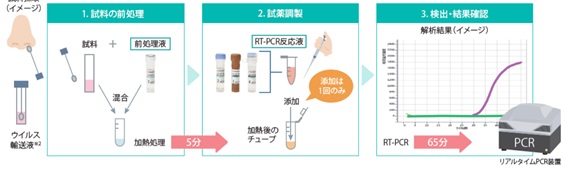
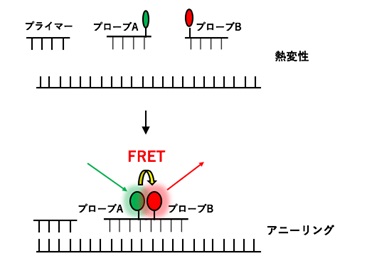
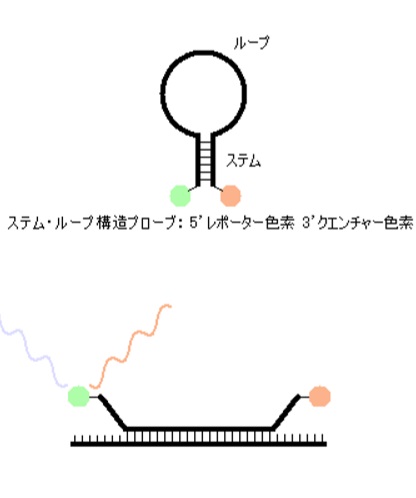
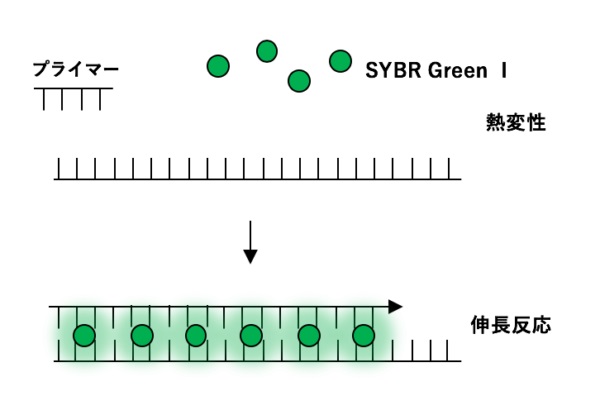
新型コロナウイルスの検査方法には、PCR検査、抗原検査、抗体検査の3種類あります。抗原検査はウイルスの持つたんぱく質、抗体検査はウイルスに対抗して生成された抗体を検出します。PCR検査はウイルスの遺伝子情報用いてウイルスを検出する検査方法です。新型コロナウイルス情報が毎日報道されるので、PCRはすっかり有名になりました。
PCR(=Polymerase Chain Reaction)法は、1983年に米国のキャリー・マリス(Kary Mullis)によって発明され、シータス社によって発展させられたDNAの増幅方法です。DNAポリメラーゼと呼ばれる酵素の働きを利用して、一連の温度変化のサイクルを経て任意の遺伝子領域を検出・検査できる量まで複製増幅する技術です。DNAポリメラーゼは、1本鎖のDNAを元の2本鎖のDNAに修復する酵素です。これは1958年コーンバーグ(A.Kornberg)らにより大腸菌から発見されました。PCRにより培養で増やせない菌やウイルスの種類を知ることができます。なお、新型コロナウイルスの検体の検査にはバイオセーフティレベル2(BSL2)の安全キャビネットと実験室と防護が必要です。作業前後にはUV灯の点灯、次亜塩素酸ナトリウム液やDNAZapもしくはDNA AWAYなどでの内部清拭が必須です。
新型コロナウイルスはRNA型のウイルスです。RNAは増幅できないので、一旦RNAを鋳型に逆転写(Reverse Transcription)して相補的なcDNAを生成させてPCR法で増幅します(cはcomplimentary)。これをRT-PCR法といいます。RTは室温でもリアルタイムのことでもないので、注意が必要です。
1991年にTW. MyersとDH. Gelfandらにより、逆転写酵素活性とDNA合成酵素活性を併せ持つ酵素(Tth DNA Polymerase)が発見され、この酵素と反応条件の工夫により1種類の試薬と1本の反応容器で逆転写反応とPCR反応が連続的に行えるようになりました。 60℃ 30分間の加温により逆転写反応は進行し、その後PCR反応に移行します。
PCR反応液の作り方を説明します。反応液は、増幅するDNAサンプル、プライマー、DNAポリメラーゼ、合成するDNAの素材である遊離ヌクレオチド(dNTP=デオキシ・ヌクレオチド三リン酸)、Mg2+(2mMのMgCl)を含むバッファー溶液(pH7.5~9.5)を混合して作製します。DNAポリメラーゼには高温で動作するTaqポリメラーゼを使います。Taqポリメラーゼはサーマスアクアティカス(Thermus-aquaticus)という好熱菌由来の酵素です。
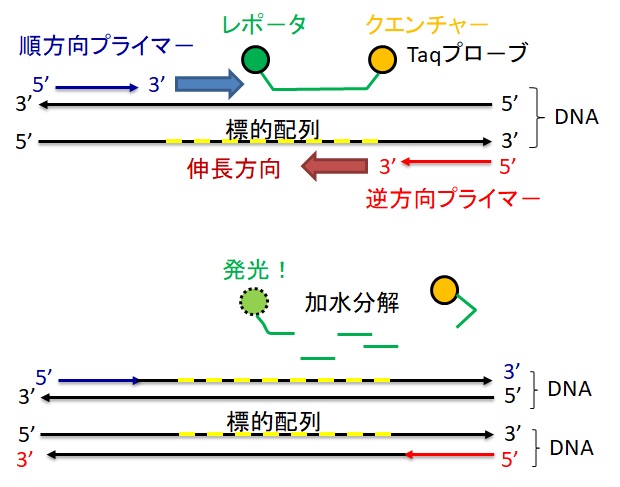
プライマーは標的DNAの一部に対して相補的な塩基配列を持つ短い一本鎖DNAです。PCRの目的は、ターゲットDNA鎖全体の複製ではなく、対象となる生命体に特有な約100~35,000塩基対のターゲット配列を複製することです。プライマーはこのターゲット配列の両端を定義する役割を持っています。
一般的に、プライマーは20~30塩基の長さからなる1本鎖の合成DNAです。塩基は4種類あるので、16mer(monomeric unit)(16塩基の長さ)のプライマ-の種類は40億(=416=4・230)にもなります。例えばランダムな30億塩基対(ヒトゲノム)のDNAに16merの長さのプライマと同じ配列は1か所にしかないと考えられます。
DNA合成は常にプライマーの3’末端から始まり、1本鎖DNAテンプレートの5’から3’方向へのみ伸長していきます。順方向プライマーと逆方向プライマーを開始点とし、相補的な2本鎖のDNA分子が効率よく合成されます。
PCRサイクルを説明します。PCRは3ステップを1サイクルとして数十回繰り返して、必要な量まで増幅します。第1ステップでは、反応液を94°Cに加熱し、30秒~1分間温度を保ち、2本鎖DNAを変性(Denaturation)させ1本鎖に分かれさせます。塩基間の結合は水素結合であり93℃程度で壊れます。変性が起こる温度は、DNAの塩基構成および長さ(塩基数)によって異なり、一般に長いDNAほど変性温度を高くする必要があります。
第2ステップでは、60℃にまで急速冷却し、1本鎖DNAとそれに結合する短い一本鎖DNAであるプライマーを結合(Annealing)させます。二本鎖DNAの50%が解離して一本鎖となる温度はTm値(melting temperature)と呼ばれています。Tm値は塩基配列の構成と塩基数および反応液の塩濃度などにより決まります。プライマーのTm値の計算には、「Nearest Neighbor法」を用いた計算ソフトがあります。アニーリング温度はプライマーのTm値(65℃)以下に設定します。アニーリング温度がプライマーのTm値以上だとプライマ-が1本鎖DNAに結合しません。アニーリング温度が低すぎると、プライマ-同志が結合してしまいます。プライマーは凍結融解を繰り返すと分解する恐れもあるので、10回程度で使い切るくらいの量に小分けして-20℃で保存します。
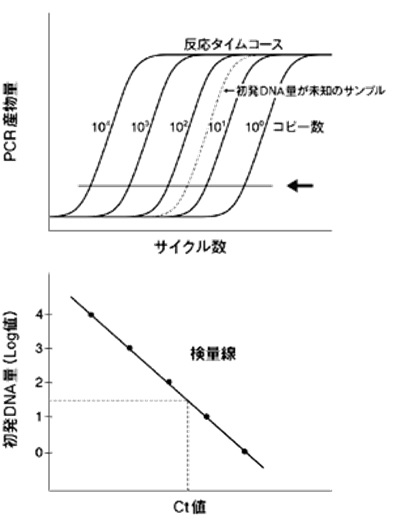
第3ステップでは、再び72°Cに加熱して、1〜2分保ちます。酵素は1分間に2~4kbを合成するので、標的配列の鎖長が1kb以下の場合は1分間で充分です。この時2つの1本鎖DNAに結合したプライマ-の隣に反応液中の塩基が次々に結合して伸長(Elongation、Extension)し、2つの2本鎖DNAが得られます。72°Cは、プライマーの分離がおきず、DNAポリメラーゼの活性に至適な温度です。わずか20サイクルのPCRにより、ターゲットのおよそ100万(220)コピーが合成されることになります。通常25~40サイクル行います。
サーマルサイクラーは、サーマルブロックと呼ばれる金属板でプログラム通りに反応チューブを急速に加熱・冷却する機能を持ちます。金属板にはヒーターやペルチェ素子などがついており的確に反応液の温度を上下させます。
PCRでDNAを増幅していくと、いずれPCRの基質(dNTP)やプライマーの枯渇などにより、温度サイクルを増やしてもDNAが増えなくなります(プラトー現象)。そのため初期のDNA濃度に数倍の差があったとしても、数十回のPCRサイクル後には増幅産物の量に差が見られなくなるため、初期DNA量の定量を行うことはできません。