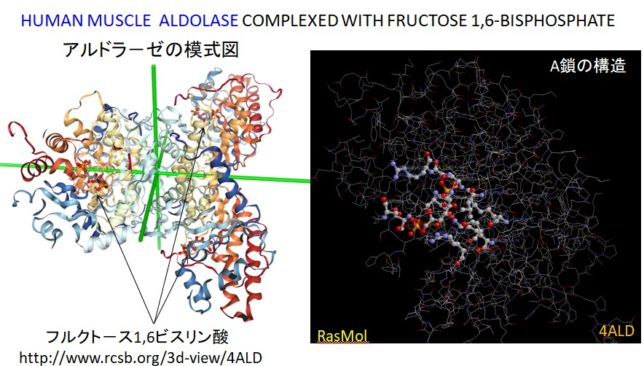
解糖系の第4ステップの酵素は、フルクト-ス・1.6ビス・リン酸(=FBP)を2つのC3化合物に分解するアルドラーゼ(Aldolase)です。タンパク質デ-タバンク(PDB)でこの酵素の構造について調べ、反応機構を推測しました。
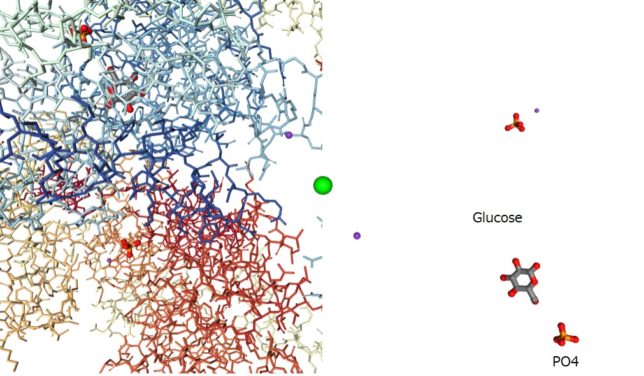
PDB番号4ALD(1998年)にヒトの筋肉の解糖系のアルドラ-ゼ酵素(FBPを含む)のデ-タがありました。ヒトの筋肉のアルドラ-ゼは363個のアミノ酸からなるタンパク質です(EC4.1.2.13)。
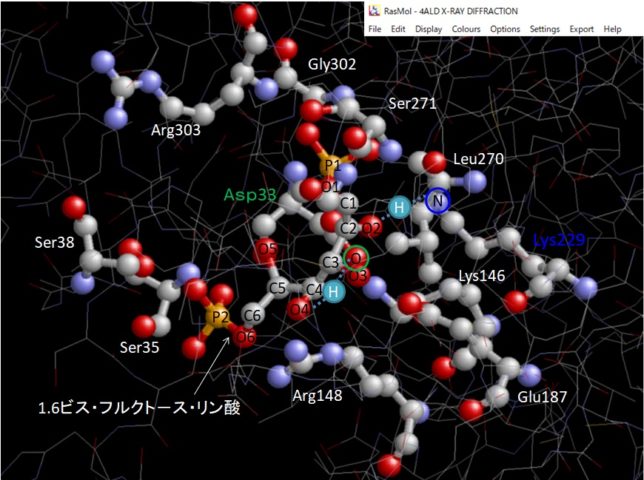
今回はRasMolという無料ソフトウエアで、タンパク質デ-タを描画し、周囲のアミノ酸を確認しました。例えばLys229を表示する場合、コマンドラインでRasMol>Select [Lys]229:Aと打ち込んで選択し、メニュ-でstick&Ballを選んでLys229を表示させます。
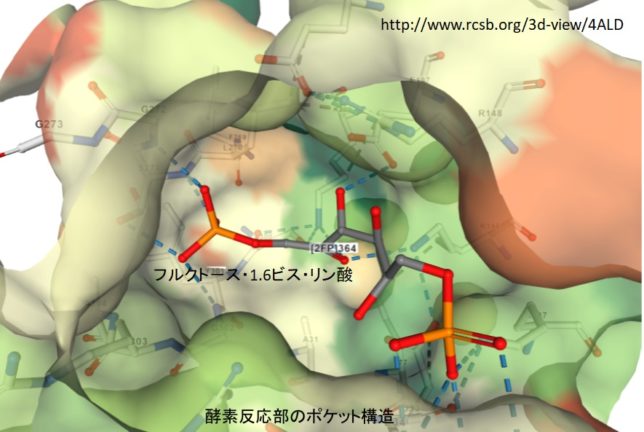
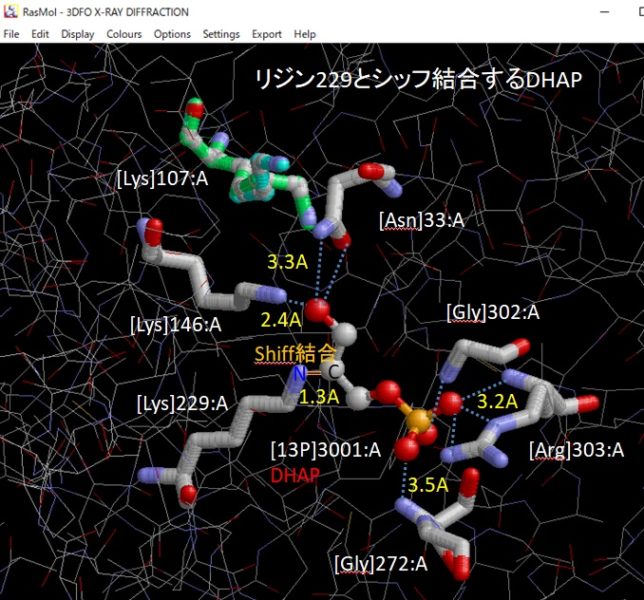
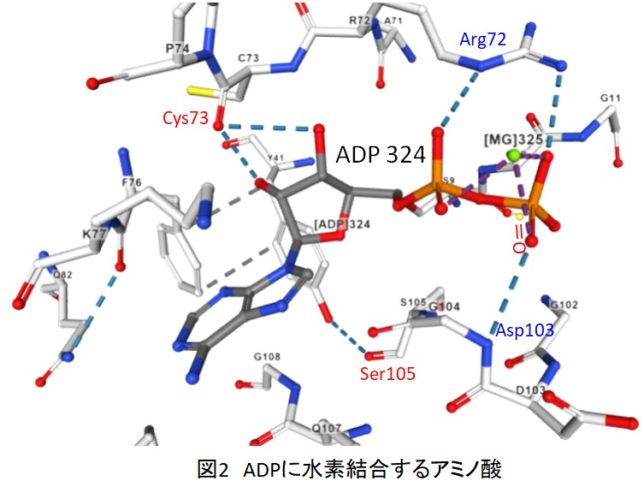
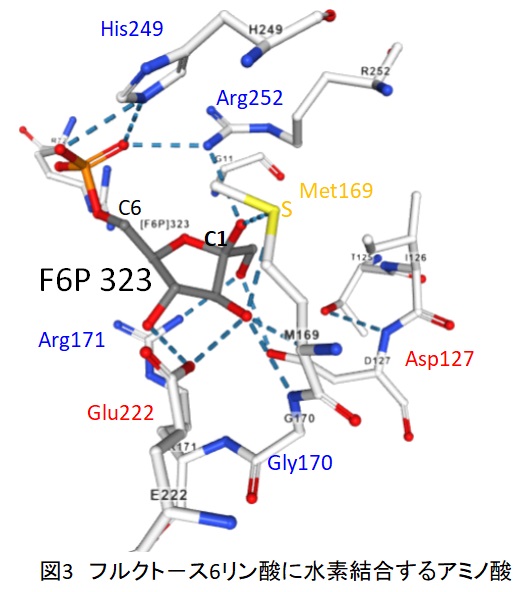
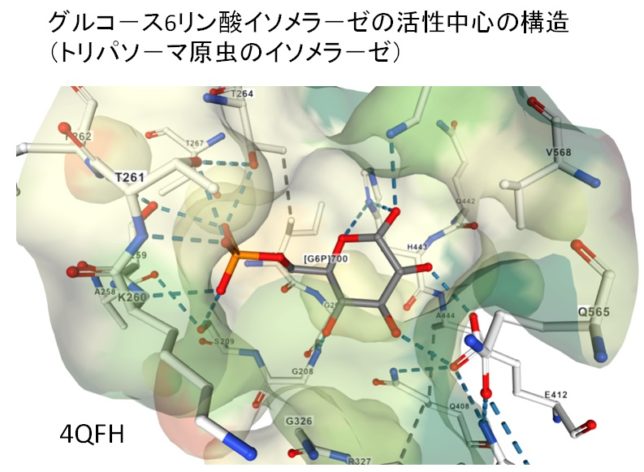
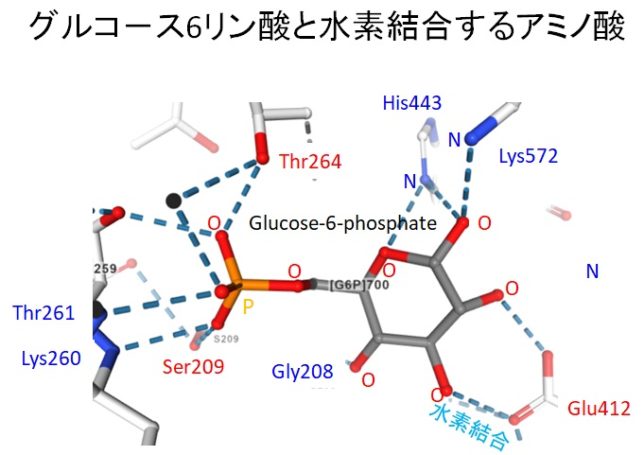
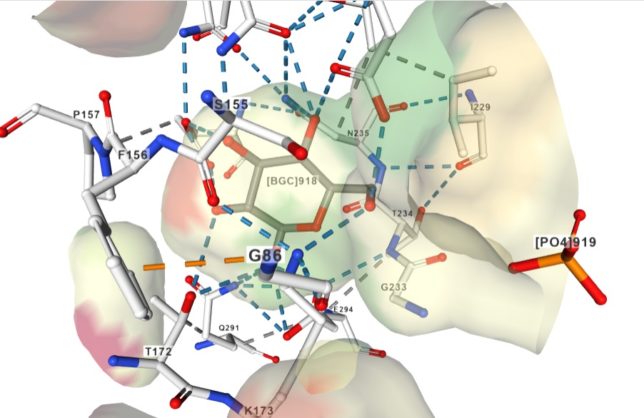
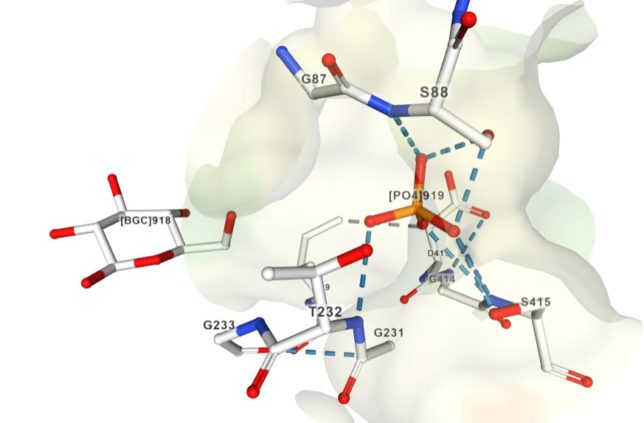
FBPフルクト-スは、酵素の反応ポケットに取り込まれ、10個程度のアミノ酸で取り囲まれ、水素結合で固定されています。229番目のリジンと33番のアスパラギン酸の触媒作用によって、FBPは2つのC3化合物に分解すると考えられます。
2つのC3化合物とは、ジヒドロキシ・アセトンリン酸(=DHAP)とグリセル・アルデヒト3リン酸(=GAP)のことです。こうした化合物の名前は、慣れるまで、発音するのも難しいです。
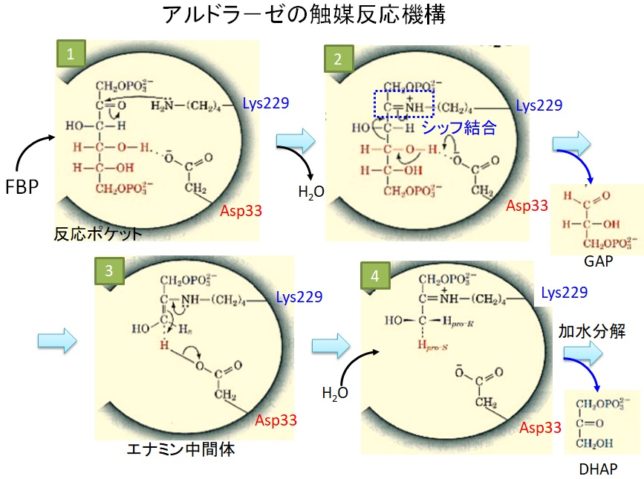
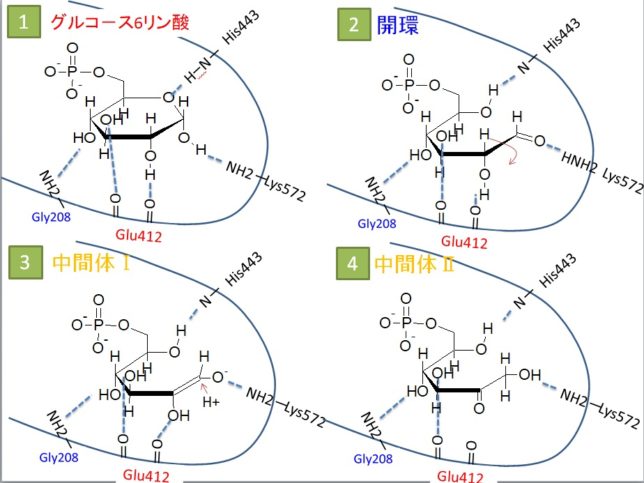
下図に示すように、酵素反応は4段階からなると考えられます。
最初にアルドラーゼに嵌まり込んだBFPフルクト-スは、開環し、FBPのC2炭素のカルボニル基(C=O)がリジン229のアミノ基(NH2)と脱水反応し、シッフ結合します。その結果C2炭素はC=OからC=N+Hのイミン状態に変化し、リジンと結合します。
私たちの血管が老化するのは、血管壁のコラ-ゲン・ペプチドが糖と反応してシッフ結合を形成するからだと思います。そういう意味でもこれは健康に関わりの深い反応です。
次にアスパラギン酸33のカルボキシル基(COO-)がBFPフルクト-スのC4炭素のOH基からH+を奪います。それによってC4炭素に電子が供給され、C3とC4の間の結合がアルド-ル開裂し、グリセルアルデヒト3リン酸(GAP)が生成れ、ポケットから離脱します。
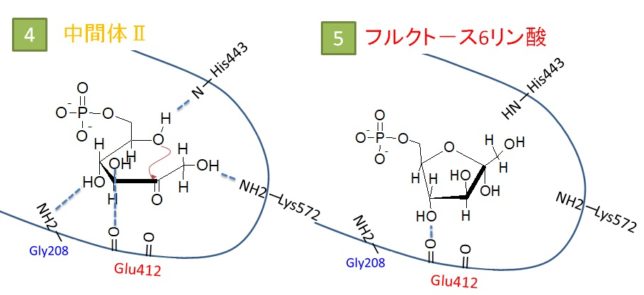
ポケットに残った化合物はエナミン状態になっていますが、アスパラギン酸のCOOH基からHを供給されると、C2炭素は元のイミン状態(C=N+H)に戻ります。最後に加水分解により、シッフ結合が解けて、C2炭素はC=Oに戻り、グリセル・アルデヒト3リン酸(GAP)となって、反応ポケットから離脱します。
次の第5ステップではDHAPはGAPに変化します。つまり1分子のグルコ-スから2分子のGAPができます。フルクト-スを分解することで、後半の反応が1本化できます。もしグルコ-スを分解すると、G2化合物とG4化合物となってしまい、後半の反応が1本化できません。
これで解糖系の前半は終了します。前半はグルコ-ス1分子に対して2分子のATPを消費しました。後半では4分子のATPを生産します。結局、解糖系全体では2分子のATPが得られます。解糖系は殆どすべての生物に見られる古い代謝系です。